10.3 配对检验和干预队列的处理方案
在涉及干预的研究中,常常需要分析同一批受试者在干预前后的组学数据(例如:溃疡性结肠炎患者在益生菌干预前和干预后肠道菌群的比较),并进行配对检验。为了确保配对检验能正常运行,需要在表型数据中将设置样本与受试者的相关信息。
🏷️示例:
Step1: 处理样本相关数据
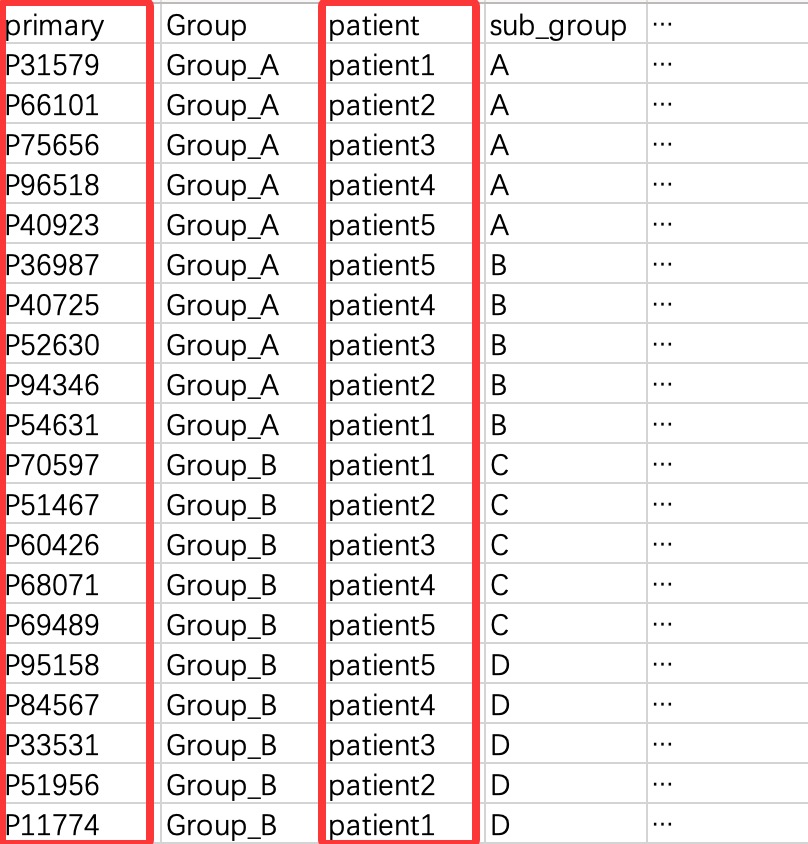
为了符合配对检验的需求,需要添加primary列的对应的配对信息。
Step2: 配对差异性检验
注意:
差异分析模块只能支持2组的配对检验,如需要多组可以直接在可视化模块中进行配对检验。
差异分析模块只能支持2组的配对检验,如需要多组可以直接在可视化模块中进行配对检验。
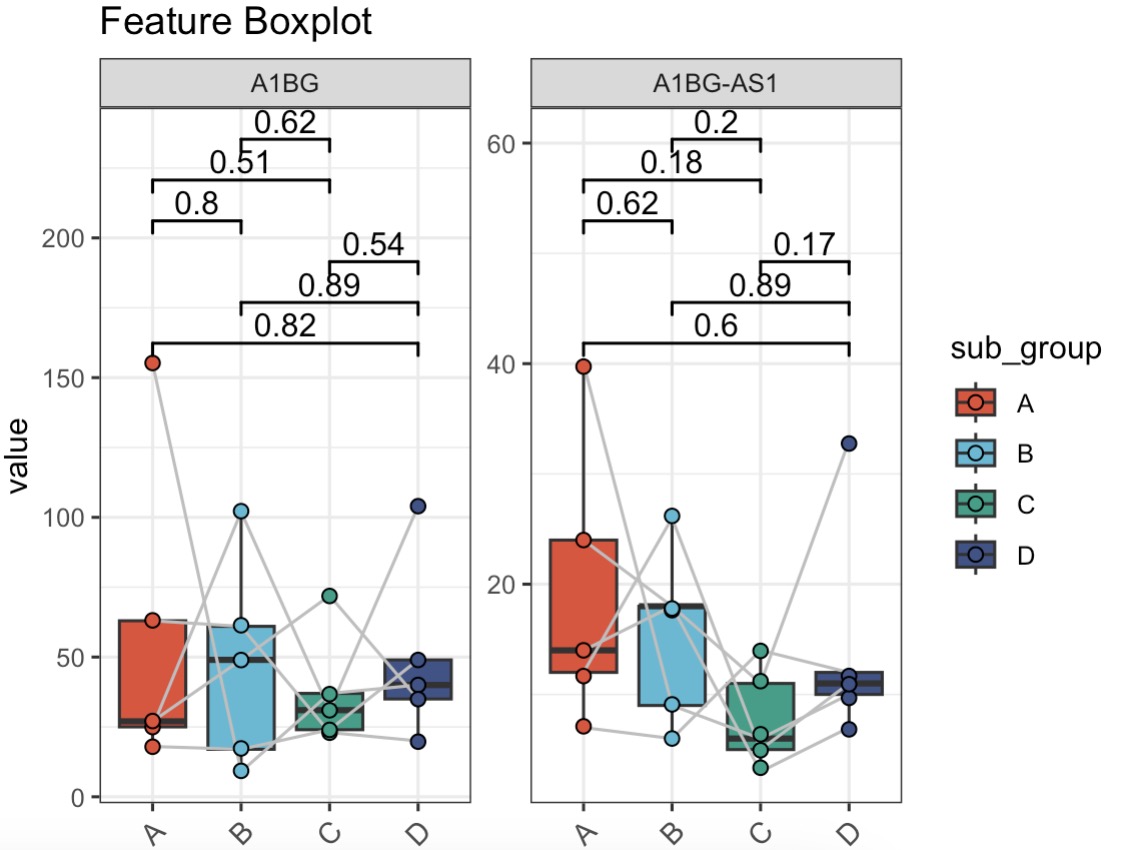
MAE |>
EMP_assay_extract('host_gene',
pattern = 'A1BG',pattern_ref = 'feature') |>
EMP_filter(sub_group %in% c('A','B')) |>
EMP_diff_analysis(method = 't.test',
paired_group='patient', # Set the paired group
estimate_group = 'sub_group')

Step3: 配对检验可视化
注意:
参数
参数
paired_line可以选择是否显示配对连线。MAE |>
EMP_assay_extract('host_gene',
pattern = 'A1BG',pattern_ref = 'feature') |>
EMP_boxplot(method='t.test',
estimate_group='sub_group',
paired_group='patient') # Set the paired_group
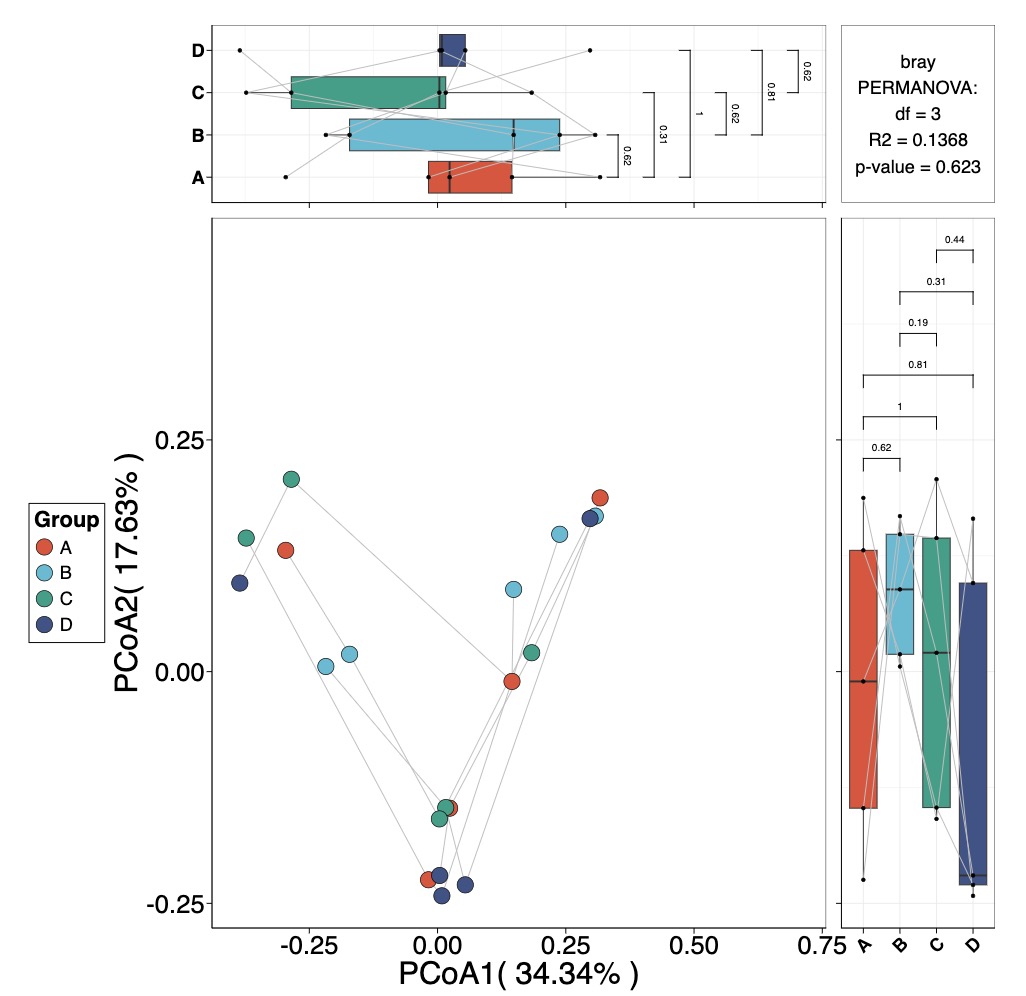
MAE |>
EMP_assay_extract('taxonomy') |>
EMP_collapse(estimate_group = 'Species',collapse_by = 'row') |>
EMP_dimension_analysis(method = 'pcoa',distance = 'bray')|>
EMP_scatterplot(show='p12html',
paired_group='patient',
estimate_group = 'sub_group')